
NEWS
新闻中心
SERVERS
服务项目
NEWS
最新资讯
发布时间:2024-05-22

编者按
线粒体脱氧核糖核酸缺失综合征(MDS)是一种严重的、表型异质的隐性遗传病,绝大多数DGUOK突变的MDS患者铁负荷过重,进展为严重的肝功能衰竭。然而,铁超载和肝损伤之间的病理机制仍不清楚。
今天,我们回顾一项由中科院广州生物医药与健康研究所刘兴国研究员团队于2021年8月发表在《Advanced Science》的经典研究——《A Combined Model of Human iPSC‐Derived Liver Organoids and Hepatocytes Reveals Ferroptosis in DGUOK Mutant mtDNA Depletion Syndrome》,该研究揭示了铁死亡的病理机制,为线粒体DNA耗竭综合征(MDS)的病理机制和治疗策略提供了重要的启示。同时,本研究还建立了首个高纯度肝细胞类器官 (iHep-Orgs)模型,为遗传性肝病的研究提供了新的工具。
文章题目:A Combined Model of Human iPSC‐Derived Liver Organoids and Hepatocytes Reveals Ferroptosis in DGUOK Mutant mtDNA Depletion Syndrome
杂志:《Advanced Science》(IF=15.1)
发表时间:2021年5月19日
作者:刘兴国研究员团队
单位:中科院广州生物医药与健康研究所等
01、研究背景
线粒体DNA耗竭综合征(MDS)是一组严重的隐性遗传疾病,其特征是组织和类器官中的mtDNA含量显著减少。脱氧核糖核苷激酶(DGUOK)的突变是导致肝功能衰竭等严重临床表现的主要原因之一,而目前除了肝移植外,缺乏有效的治疗方法。铁超载与DGUOK突变MDS患者的肝功能衰竭密切相关,但其病理机制尚未完全揭示。
为了深入研究其内在机制并进行药物发现,建立一个有效的模拟DGUOK突变MDS患者肝脏的模型系统至关重要。作者提出了使用iPSCs诱导出的肝类器官和肝细胞是一个很好的模型,这些模型可以提供患者的完整的遗传背景,并且具有研究潜力。
作者还建立了一种体外肝脏疾病模型,使用DGUOK突变患者的iPSCs诱导出肝类器官和肝细胞,并证明了这些细胞对铁超载诱导的铁死亡更为敏感,同时也提供了针对这一过程的潜在治疗策略。
02、研究结果
1. 从CRISPR/cas9介导的基因校正DGUOK突变iPSC生成肝脏类器官和肝细胞模型
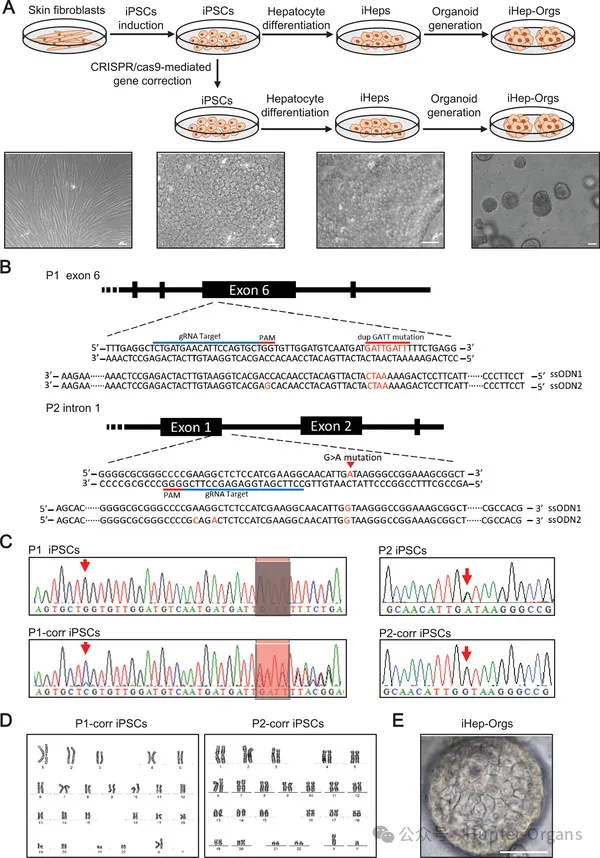
原文图1 CRISPR-cas9介导的基因中生成肝细胞和类器官模型,校正DGUOK突变iPSC
作者使用两位DGUOK基因不同突变位点的患者的皮肤成纤维细胞重新编程成iPSCs。然后,对这些iPSCs进行基因编辑,修复了患者的DGUOK基因突变。紧接着,针对对照iPSCs、患者iPSCs和修复后的iPSCs进行了肝细胞(iHep)的分化。最后,使用这些iHep产生了肝脏类器官(iHep-Orgs)。
作者还对这些iHep和iHep-Orgs进行了多种功能性检测,包括对肝脏发育和成熟的标志物的表达检测,过碘酸雪夫染色(PAS)染色和吲哚菁绿(ICG)摄取和释放等。
2. iHep-Orgs的鉴定和功能表征
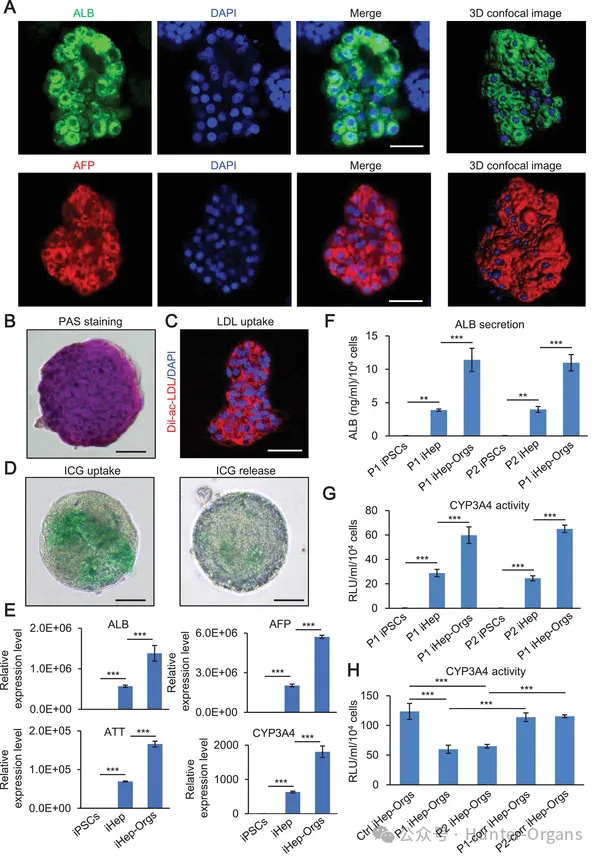
原文图2 iHep-Orgs的表征
作者通过免疫荧光、功能性染色和基因表达分析,发现iHep‐Orgs在3D培养中表现出比2D培养中的iHep更为成熟和功能性强的肝脏特性。
与此同时,对患者iHep‐Orgs进行的检测显示部分异常的肝功能,特别是CYP3A4活性下降,但ALB分泌未受影响。这些结果表明iHep‐Orgs在模拟疾病相关的肝功能异常方面具有潜在的应用前景。
3. iHep患者的线粒体DNA耗竭和呼吸功能障碍
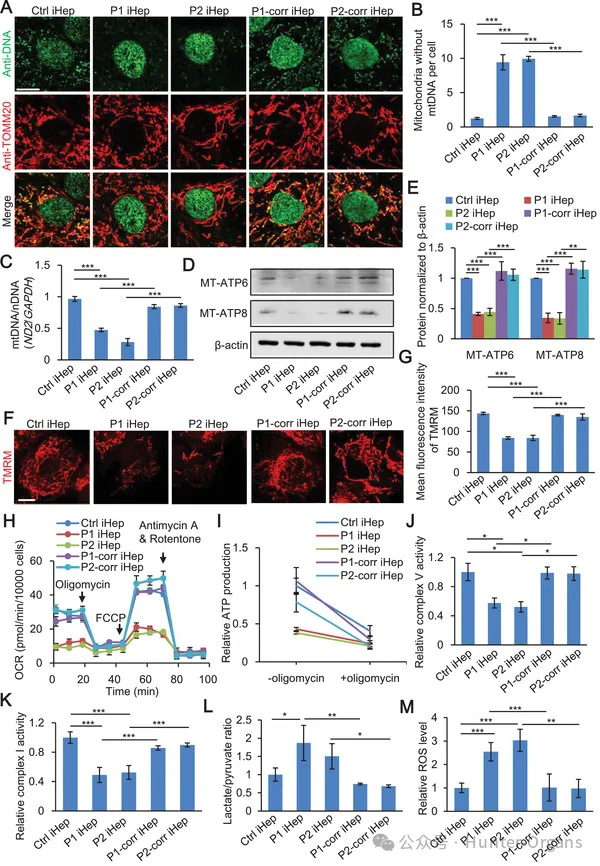
原文图3 患者 iHep 的线粒体 DNA 耗竭和呼吸功能障碍
作者通过免疫荧光和荧光定量PCR检测发现,患者iHep中的线粒体DNA(mtDNA)含量显著减少,与对照组和DGUOK矫正iHep相比存在明显的差异。
进一步的研究发现,患者iHep的线粒体RNA和蛋白质水平也显著降低。在评估线粒体生物能的过程中,通过荧光染色和耗氧率检测发现,患者iHep的线粒体膜电位、基础耗氧率和最大呼吸率均明显降低,这表明其线粒体呼吸功能受损。
此外,患者iHep的细胞和线粒体ATP产生能力也显著减少,线粒体呼吸链复合物的活性也受到影响。最后,患者iHep中的乳酸/丙酮比和ROS水平均显著升高,进一步证明了其线粒体功能异常。这些结果强调了DGUOK突变患者肝细胞中线粒体功能受损的重要性。
4. GSH前体NAC挽救了患者iHep-Orgs和iHep对铁过载诱导的铁死亡的敏感性
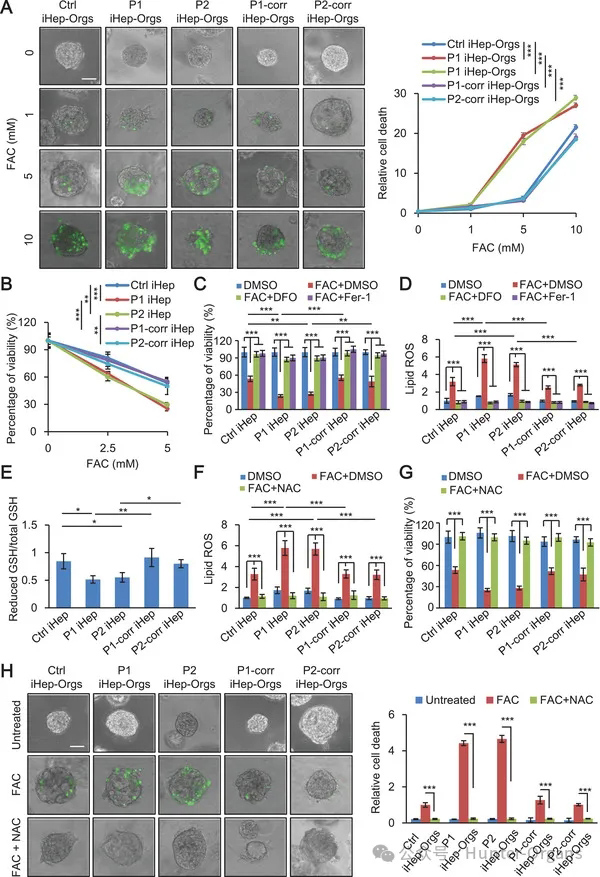
原文图4 GSH前体NAC挽救了患者iHep-Orgs和iHep对铁超负荷诱导的铁死亡的敏感性
作者发现患者iHep和iHep‐Orgs中转铁蛋白(TF)的表达增加,表明肝细胞中的铁载体转铁蛋白的表达水平升高。紧接着,通过给予铁过载剂柠檬酸铁铵(FAC)处理,发现患者iHep‐Orgs和iHep对铁过载诱导的细胞死亡更为敏感。
进一步的实验显示,使用特异的铁死亡抑制剂去铁灭亡抑制剂(DFO)和铁抑素-1(Fer-1)能够抑制铁过载诱导的细胞死亡,表明铁过载引发的细胞死亡主要是通过铁过量引起的铁死亡机制。
此外,实验还发现患者iHep中的脂质过氧化水平较高,而且谷胱甘肽 (GSH)水平下降,这导致了铁过载诱导的脂质过氧化增加和对铁死亡的敏感性。然而,通过使用GSH前体 N-乙酰半胱氨酸 (NAC)能够恢复GSH水平,减轻脂质过氧化,并且减轻了铁过载诱导的铁死亡。这些结果强调了GSH水平下降导致的脂质过氧化增加和对铁死亡的敏感性,可以通过GSH前体NAC进行干预。
5. 患者iHep的铁死亡伴随着溶酶体中铁蛋白的降解
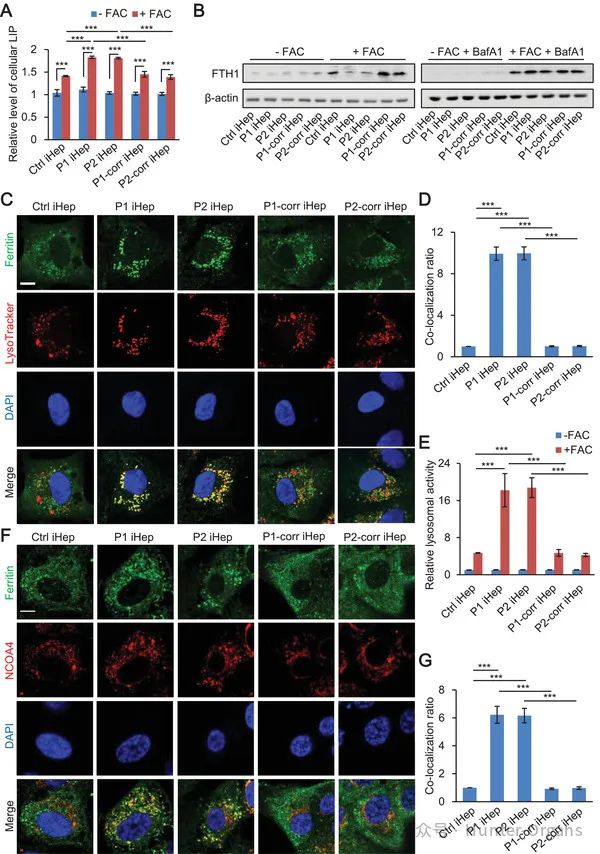
原文图5 患者iHep的铁死亡伴随着溶酶体中铁蛋白的降解
作者在铁过载的iHep中测量了可溶性铁(LIP)的水平,并发现LIP在接受5 × 10−3 m FAC处理48小时后增加,患者iHep中的LIP水平高于对照组和DGUOK修复组。
进一步研究表明,与对照组和修复组相比,患者iHep中FTH1的表达水平在FAC处理后没有显著增加。使用溶酶体酸化抑制剂BA处理时,患者iHep中FTH1的水平与对照组和修复组相似,表明FTH1在患者iHep的溶酶体中被降解。进一步的研究分析了铁蛋白与溶酶体的共定位,结果显示患者iHep中铁蛋白与溶酶体的共定位程度显著高于对照组和修复组。
另外,研究还表明,在FAC处理后,患者iHep中的溶酶体活性显著高于对照组和修复组。最后,通过免疫荧光检测,发现患者iHep中铁蛋白与核受体共激活因子4(NCOA4)的共定位比例也更高。这些结果表明,在患者iHep中,铁蛋白的溶酶体降解是铁死亡的一个步骤。
6. 患者iHep的铁死亡是溶酶体中铁蛋白 NCOA4 依赖性降解的过程
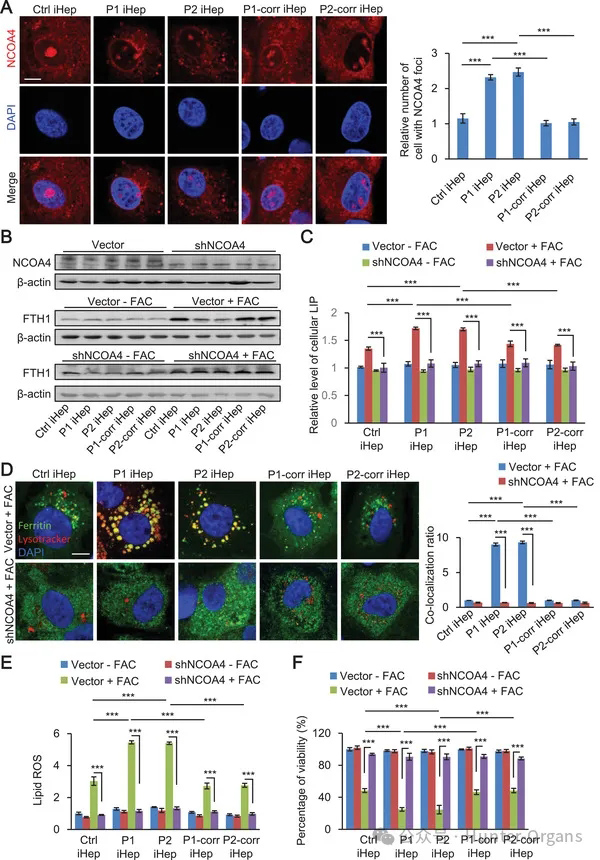
原文图6 患者iHep的铁死亡是溶酶体中铁蛋白的 NCOA4 依赖性降解过程
作者在进行FAC处理前检查了iHep中NCOA4的免疫荧光情况。令人惊讶的是,与对照组和修复组相比,患者iHep中出现了许多NCOA4焦点。
然后,作者探究了NCOA4是否是导致患者iHep铁死亡中铁蛋白降解的关键因素。作者使用短发夹RNA (shRNA) 干扰来沉默NCOA4表达,并观察到在FAC诱导的患者iHep中FTH1水平的增加。随后,作者测量了LIP水平,并发现NCOA4沉默使LIP水平降低到与受铁过载的载体处理的iHep相当的程度。
为进一步确定NCOA4在铁蛋白的溶酶体降解中的作用,作者分析了在NCOA4敲除后铁蛋白与溶酶体的共定位。结果显示,在NCOA4缺乏的情况下,铁蛋白呈现出一种弥散的定位模式,而不与溶酶体共定位。
此外,作者还发现NCOA4缺乏还阻止了铁过载条件下的脂质过氧化和铁死亡。这些结果表明,在铁过载诱导铁死亡过程中,NCOA4介导了溶酶体中铁蛋白的降解。
03、编者点评
作者揭示了DGUOK突变MDS患者iHep以及患者的iHep和iHep-Orgs的 mtDNA耗竭和呼吸功能障碍对铁过载诱导的铁死亡更敏感,而这种铁死亡可以通过谷胱甘肽前体NAC进行救治。此外,这种铁死亡是由溶酶体中铁蛋白的 NCOA4 依赖性降解介导的过程,可以通过NCOA4沉默来挽救,为研究MDS患者肝衰竭的新治疗途径奠定了基础。
作为健康美丽产业CRO服务开拓者与引领者、斑马鱼生物技术的全球领导者,环特生物搭建了“斑马鱼、类器官、哺乳动物、人体”四位一体的综合技术服务体系,开展健康美丽CRO服务、科研服务、智慧实验室搭建三大业务。目前,环特已建立200多种斑马鱼模型及人诱导多能干细胞及多种癌种的类器官培养平台,欢迎有需要的读者垂询!
参考文献
[1] Finsterer J., Ahting U., Can. J. Neurol. Sci. 2013, 40, 635.
[2] M Sezer T., Ozcay F., Balci O., Alehan F., J. Child Neurol. 2015, 30, 124.
[3] Yan N., Zhang J.‐J., Front. Neurosci. 2019, 13, 10.3389/fnins.2019.00811.
[4] Hu H., Gehart H., Artegiani B., LOpez‐Iglesias C., Dekkers F., Basak O., van Es J., Chuva de Sousa Lopes S. M., Begthel H., Korving J., van den Born M., Zou C., Quirk C., Chiriboga L., Rice C. M., Ma S., Rios A., Peters P. J., de Jong Y. P., Clevers H., Cell 2018, 175, 1591.